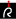 NBRP情報センター
NBRP情報センター
山崎 由紀子 (国立遺伝学研究所 系統生物研究センター 系統情報研究室)
|
今月は情報センターからナショナルバイオリソースプロジェクト(NBRP)のポータルサイトを紹介します。NBRPは2002年に発足し、2016年4月には第3期(1期5年)の最終年に入ります。NBRPは4つのプログラム:
(1)リソース整備プログラム
(2)情報センター整備プログラム
(3)ゲノム情報等整備プログラム
(4)技術基盤整備プログラム
から構成され、中心的プログラムであるリソース整備プログラムは「研究開発材料としてのバイオリソースの収集・保存・提供等の体制整備」を目的として、29のリソースグループによって進められています。2番目のプログラムは当情報センターが担当しており、「NBRPポータルサイト(www.nbrp.jp)の運営」と「リソース整備プログラムの情報公開支援(現在29のうち20のリソースの情報公開を支援)」を2大ミッションとして活動しています。 |
プロジェクトに関する情報はすべてポータルサイト(図1)から公開しています。全グループの情報公開サイトへは図1-A から夫々アクセスでき、担当機関と代表者の最新情報は図1-B から得ることができます。
上述の(3)(4)のプログラムについては図1-C,D からその内容と成果を公開しています。情報センター整備プログラムの3つの分担課題(「地球規模生物多様性情報機構の日本ノードの活動:GBIF」、「大型類人猿情報ネットワーク:GAIN」、「ABS(名古屋議定書)対応」)は、それぞれ図1-E,F,G から活動状況を公開しています。NBRPリソースを使った研究論文の情報(Resource Research Circulation:RRC)は図1-H から閲覧できます。オンライン上で登録も可能です。利用者からフィードバックされた論文情報が直ちにリソース情報に反映できるよう、RRCと各リソースデータベースとの連携も進めています。
|
|

図1. NBRP ポータルサイト |
PubMedは世界中の研究者に利用されている文献情報のデータベースですが、個々の論文に関連する情報を外部DBにリンクしてくれるLinkOutというサービスを提供しています。RRCはこのサービスを利用して9000報以上の論文をNBRPリソースの情報サイトとリンクしています。
同様に、リソース関連でLinkOutに登録しているグループには、Addgene, ATCC, CCR, GCM, JAXMice, PCC, GRINなど海外の主要なリソース機関も含まれており、登録機関の数は年々増加しています。論文に使われた研究材料を、著者から個人的に入手するのではなく、リソースセンターから、品質保証付きで確実に、かつ正式な手続きを経て入手することができる理想的な環境が整いつつあることがわかります。
ポータルサイトのもう1つの特徴は、29グループから提供されている全リソース(2016年2月現在650万件)を一括検索(BioResource World:BRW)できることです。
ポータルサイトから希望する特定のリソースの情報にアクセスできることはもちろんですが、利用者の研究目的に叶うリソースの候補をシステムから提案できるようなポータルサイトができたらよいといつも思っています。 |
|
その第一段階として、キーワード検索やDNA配列の相同性検索のほか、遺伝子オントロジーや疾病オントロジーなどからも一括検索できるようにしています(図2A)。これらのオントロジーは情報センターが外部のデータベース(PubMed MeSH, Homologene, OMIM, dictyBase, WormBase, ZFINなど)を利用して独自に付与しているものであり、まだ十分ではありませんが、少しずつオントロジー付きのリソースが増えているところです(図2B)。
|
|

図2. BRW Ontology検索
A. Gene Ontology、B. Disease Ontology |
現状は、生物種を特定してリソースを探すという利用者の方が圧倒的に多いのですが、研究目的によっては、異なる生物種の方が適しているという場合もあるでしょう。しかし新しいリソースを使う時のハードルは結構高いはずです。こうした利用者のためにNBRPの多くのリソース機関では初心者向けに取り扱いの講習会を実施して便宜を図っています。情報センターとしても、より多くの選択肢から最適な研究材料を選択できる仕組みを提供できるよう、情報を一層充実させていきたいと思っています。
|