アサガオの新規突然変異を利用した
「花の色を濃くするタンパク質」の発見
星野敦 (基礎生物学研究所・モデル生物研究センター 助教)
アサガオには、江戸時代の後期に生じた突然変異が多く現存しています。これらは、「Tpn1ファミリーのトランスポゾン」と呼ばれる動く遺伝子が、ほかの遺伝子に挿入することで生じたものです。このトランスポゾンは、アサガオのゲノム中に800ほどもあるうえに、現在でも動く活性があるので、しばしば新しい突然変異を生みます。そのような新しい突然変異を利用することで、「花の色を濃くするタンパク質」が発見されました。 |
|
花の多くは、アントシアニンにより彩られています。アントシアニンは植物の代表的な色素で、フラボノイドと呼ばれるポリフェフェノールの一種です。花の色の濃さは、そこに含まれるアントシアニンの量によって決まり、多く含まれるほど色が濃くなります。これまで、アントシアニンの含有量を調節する因子や仕組みについての知見は限られていました。 |
今回の研究では、アントシアニンの含有量を少なくして花の色を薄くする性質をもつ、新しい変異が使われました。この変異は人工的に作られたものではなく、「紅ちどり」という市販の系統から自然に現れました。実際には、紅ちどりから直に現れたのではなく、3度も突然変異が繰り返しておきて現れました(図1)。 |
|
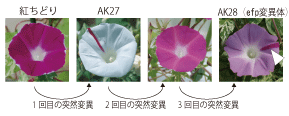
図1:AK28の薄いピンク色の花には、濃い赤色の斑点(白い矢の先など)が観察されます。 |
|
はじめの変異では、濃い赤色の花の紅ちどりが、白地に赤い斑点模様の花を咲かせるようになりました。この変異は、平成12年に富山県の一般の方が見つけました。その種子を私たちが入手して栽培していたところ、さらに2度の突然変異がおきました。1つは、富山で現れた突然変異が復帰変異をおこして、もとの赤い花を咲かせるようになるもの。もう1つは、この研究に利用された花の色を薄くするという変異でした。このように偶然が重なって得られた薄い赤色の花を咲かせる系統(AK28)は、新しいリソースとしてNBRPアサガオによって収集されました。
|
AK28の薄い赤色の花には、紅ちどり本来の濃い赤色の斑点模様が観察されます。また、AK28の種をまくと、紅ちどりと同じように濃い赤色の花を咲かせる復帰変異体も得られます。このような表現型は、トランスポゾンが関与する変異によく見られるものです。そこで、アサガオのTpn1ファミリーのトランスポゾンが突然変異をおこしたと仮定して原因遺伝子を探索しました。探索には「トランスポゾン・ディスプレー法」という、トランスポゾンに接したDNA配列をPCRで増幅する方法を用いました(図2)。変異体ではトランスポゾンが挿入しているので原因遺伝子に由来するDNA断片が増幅するが、野生型の植物や復帰変異体では増幅しないという原理にもとづく方法です。この方法で見つかった原因遺伝子は、それまで機能が知られていないタンパク質をコードしていました。花の色素を調べると、アントシアニンだけでなく、無色のフラボノールというフラボノイドの生合成も促進する機能を持つことが分かったので、Enhancer of Flavonoid Production(EFP)と名付けました。EFP は、EFPが働かない場合に比べてアントシアンの生産効率を3倍程度に高めています。
さらに私たちは、NBRPアサガオで収集されている薄い色の花の39系統について、EFP遺伝子を調べました。その結果、そのうち32系統は機能欠損したEFP遺伝子をもつことが分かりました。また、ペチュニアやトレニアのEFPも、アサガオの場合とおなじようにフラボノイドの生産効率を高める働きがあることが明らかになりました。
|
| |
|
|
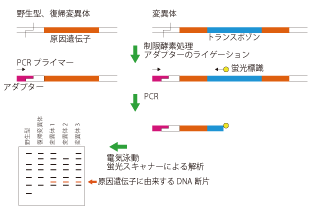
図2:トランスポゾン・ディスプレー法。
変異体では、原因遺伝子にトランスポゾンが挿入しています。ゲノムDNAを制限酵素で切断し、切断部分にアダプターを結合させたあと、アダプターとトランスポゾンの配列上に設計したプライマーを用いてPCRを行います。トランスポゾン側のプライマーは蛍光標識してあるため、電気泳動像を蛍光スキャナーで解析すると、このプライマーにより増幅したDNA断片だけが検出されます(アダプター側のプライマー同士で増幅したDNA断片は検出されない)。変異体に共通して現れる原因遺伝子に由来する断片を、ゲルから回収して塩基配列を決定するなどして、原因遺伝子を特定します。 |
|
今のところEFPが、どのような仕組みでフラボノイドの生産効率を高めているのかは謎のままです。これを解き明かすことで、植物が物質生産を効率化する仕組みを理解できると考えています。また、本研究のように、アサガオではトランスポゾンを利用して新しい突然変異とその原因遺伝子を見つけることができます。私たちは、トランスポゾンを利用した新しい突然変異体の選抜(スクリーニング)も進めています。これまでにない科学的価値や鑑賞価値の高い変異体が得られることを期待しています。
|
| |
参考文献 |
| |
Morita, Y, Takagi, K, Fukuchi-Mizutani, M, Ishiguro, K, Tanaka, Y, Nitasaka, E, Nakayama, M, Saito, N, Kagami, T, Hoshino, A, Iida, S. (2014) A chalcone isomerase-like protein enhances flavonoid production and flower pigmentation. The Plant Journal 78, 294-304. Doi:10.1111/tpj.12469
|