Kaede蛍光タンパク質を利用したカタユウレイボヤ(Ciona intestinalis)の神経系の研究
堀江 健生1・笹倉 靖徳1, 2
筑波大学 下田臨海実験センター1, 准教授2
|
カタユウレイボヤ(写真1)を含むホヤのグループは脊索動物門に属する動物で、その幼生は典型的なオタマジャクシ型の体制を取り(写真1A)、脊索や背側中枢神経系といった脊椎動物と共通のボディプラン*1 を備えています。多くのホヤの発生は単純で、例えば初期胚や幼生は数えられる程度の細胞から構成されており、細胞系譜*2 が明らかとされています。さらにホヤは、卵のそれぞれの領域が将来の体のどの部分になるかが発生初期に決められている「モザイク卵」であるため、発生の研究においても注目されている動物です。 |
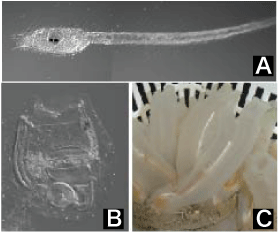 |
写真1: ホヤの一種 カタユウレイボヤ
(A) カタユウレイボヤの幼生
幼生の時期はオタマジャクシ型の体制を取り活発に遊泳する。
(B) 変態直後のカタユウレイボヤ
変態後は尾部を失い固着生活を送る。
(C) カタユウレイボヤ成体 |
|
カタユウレイボヤは、ホヤの中でも遺伝子機能解明に適した研究材料です。ゲノム配列は2002年に決定され1)、ゲノムサイズは半数体辺り約160Mbpで、遺伝子をおよそ15,852個含んでいると推定されました。この数値から、カタユウレイボヤが非常にコンパクトなゲノムを有していることが分かります。また、カタユウレイボヤにおいては卵への顕微注入による外来DNAやRNAの導入による遺伝子機能解析法が盛んに進められています。特に遺伝子の機能阻害法としてはモルフォリノオリゴ*3 の顕微注入による翻訳阻害が利用可能です。さらに、外来DNAを何百もの卵や初期胚にエレクトロポレーション法(電気穿孔法)により一度に導入ことも可能です。このように、遺伝子の配列情報を入手でき、遺伝子機能解析の各種実験技術が確立しているという特徴から、カタユウレイボヤは脊索動物のモデル生物とも言われています。
|
|
カタユウレイボヤ・トランスジェニック系統の作製
カタユウレイボヤのもう一つの特徴は、世代時間が2-3ヶ月と比較的短いことです。この短さに着目し、我々の研究グループではこのホヤの遺伝学的技術基盤を整備することを進めてきました。まず、室内閉鎖飼育系を構築しました。これにより、カタユウレイボヤを研究室内で飼育し子孫を得ることや、遺伝子組換え体を封じ込めて飼育すること、つまり遺伝学的な実験を行うことが可能になりました。続いてトランスジェニック系統*4 の作製を進めることにしました。我々は、Tc1/mariner スーパーファミリーのトランスポゾン*5 の1つMinos がこのホヤで転移活性を有していることを明らかにし2)、Minos をカタユウレイボヤゲノムへと挿入させたトランスジェニック系統を作製することに成功しました(図1)。 |
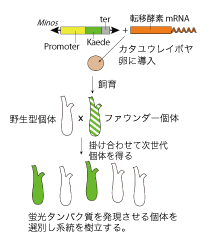 |
図1: Minos トランスポゾンによるカタユウレイボヤの形質転換
レポーター遺伝子*6を組み込んだMinos ベクターを転移酵素mRNAと共にホヤ卵に導入する。導入された個体の一部はMinos 挿入を次世代に伝える。
|
|
Minos トランスポゾンベクターと転移酵素*7mRNAを初期発生の時期に導入した個体を飼育し、その子孫を得ますと、およそ30%程度の個体がMinos 挿入を子孫に伝えます。つまり、Minos がホヤゲノムに挿入され、それが遺伝することを意味しています。最近では転移酵素を生殖細胞で発現させる系統が作製され、この系統とMinos の挿入を持つ系統を掛け合わせることにより、生殖細胞のゲノム中でMinos が転移酵素の働きで別の領域へと転移し、新しい挿入を持つ系統を作製できる系も構築されました3)。さらに、Minos を用いてゲノム中の転写制御領域であるエンハンサー活性を検出するエンハンサートラップ法*8や4)、Minos の挿入により遺伝子を破壊する挿入突然変異体の作製にも成功しています5)。Minos を用いて作製されたトランスジェニック系統の多くは、ナショナルバイオリソースプロジェクト(NBRP)に寄託されており、提供体制が整えられています。
| *1 |
生物の体をつくる設計図 |
| *2 |
受精卵から成体ができるまでの各細胞の分化の道筋 |
| *3 |
モルフォリノオリゴヌクレオチド。人工的な核酸類似物質。遺伝子の研究では主にmRNAの翻訳開始点付近に相補的な塩基配列を持つモルフォリノオリゴが作製され、そのオリゴを細胞に注入するとmRNAに結合して翻訳を阻害することができる。 |
| *4 |
その生物が本来は有しない、外来性のDNAをもつ系統 |
| *5 |
生物のゲノムDNA中を転移する性質を持つDNAのこと。末端に繰り返し配列を持つなどの特徴がある |
| *6 |
特定の生命現象を簡便に検出するために用いられる遺伝子の総称 |
| *7 |
トランスポゾンの転移に必須の働きをする酵素 |
| *8 |
レポーター遺伝子がエンハンサー活性のあるゲノムDNAの近傍に挿入された時レポーター遺伝子の発現がそのエンハンサーにより制御されて変化することを利用して、ゲノム中のエンハンサーを検出する方法
|
|
|
Kaedeを発現するカタユウレイボヤ系統
Kaedeはサンゴから単離された蛍光タンパク質6)で、紫外光によって色が緑から赤に変換する特性(光変換活性)を有しています。オワンクラゲ由来の緑色蛍光タンパク質も細胞を光らせることができますが、Kaede蛍光タンパク質の場合、光交換活性を利用して細胞の位置や時期をより細かく指定してマーキングし、細胞のその後の変化を追跡することができるのです。
このKaedeを神経系全体や特定のニューロン、特にコリン作動性ニューロン、GABA /グリシン作動性ニューロン、グルタミン酸作動性ニューロン、ドーパミン作動性ニューロンで発現させたカタユウレイボヤ系統(写真2)が作製され、ナショナルバイオリソースに寄託されています。ホヤの幼生は100個程度のニューロンから構成される単純な神経系を有していますので、これらのKaede系統は、全神経ネットワークを徹底的に研究する上で強力なマーカーとして期待されています。 |
 |
写真2: Kaedeを幼生の神経系で発現させるトランスジェニック系統
(A) 神経系全体でKaedeを発現させる系統の幼生
(B) コリン作動性ニューロンで特異的に Kaedeを発現させる系統の幼生
|
|
|
Kaedeの光変換を利用した神経細胞の変態過程での追跡
前述のKaede系統を利用した研究を一つ紹介します。ホヤは、幼生の時期にはオタマジャクシ型の体制をとり活発に泳ぎますが、変態後は固着生活を送るようになります(写真1参照)。このように、生活スタイルまでが大きく変化するホヤの変態過程では、中枢神経系が大規模な改変を受けると考えられてきました。これまでは、ホヤ幼生の中枢神経系は一度完全に失われ、成体の神経系は変態過程で新たに形成されるというのが通説になっていました。しかしながら、ホヤ幼生の神経系の細胞を、変態過程で正確にトレースした実験はこれまでなく、幼生の神経系の細胞が消失するのか残るのかについては、議論になっていました。
このことに対して、上記のKaede系統を利用してアプローチしました。具体的には、Kaedeの光変換を利用して幼生の中枢神経系の細胞をKaedeの赤色蛍光でラベルし(写真3A)、その赤色蛍光を発する細胞が、変態後に残っているのか消失するのかを解析しました。その結果、幼生の中枢神経系由来の細胞の多くは変態後も残っており、成体の中枢神経系を構築していることが分かりました(写真3B,C)。続いて中枢神経系のどの細胞タイプが残るのかを追跡したところ、幼生のニューロンの大多数は消失すること、変態後にメインに残っているのは、幼生のグリア細胞である上衣細胞であることが分かりました(上衣細胞はホヤの中枢神経系から報告されている唯一のグリア細胞です)。 |
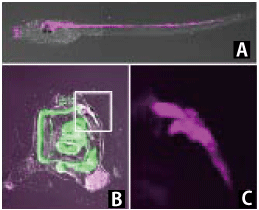 |
写真3: Kaedeを用いた幼生神経系の細胞の変態過程でのトレース実験
(A) 幼生の神経系の細胞をKaedeの光変換を利用して赤色蛍光でラベルした状態。
(B) (A)の幼生を変態後まで飼育し、赤色蛍光でラベルされた神経系の細胞が変態後の中枢神経系に残っていることを示した写真。
(C) (B)の四角い枠の部分を拡大したもの
|
|
|
さらに、上衣細胞の一部は変態中にニューロンに分化し成体ニューロンを補っていること、つまり、カタユウレイボヤの変態過程では、幼生のグリア細胞が神経幹細胞的に働き、成体ニューロンのソースとなっているということが分かりました7)。このようにKaede系統は細胞のトレース実験に強力なツールとなります。Kaedeに限らず、蛍光タンパク質で組織を特異的にラベルしたトランスジェニック系統も、発生メカニズムの解明の強力なツールになります。世界でもこのようなホヤ系統が整備されているのは、日本のNBRPリソースだけになります。その日本独自のリソースを利用して、ホヤの発生メカニズムを独創的な視点から解明したいと考えています。 |
文献
1) Dehal et al., 2002. Science 298:2157-2167.
2) Sasakura et al., 2003. PNAS 100:7726-7730.
3) Sasakura et al., 2008. Dev. Dyn. 237:39-50.
4) Awazu et al., 2004. Dev. Biol. 275:459-472.
5) Sasakura et al., 2005. PNAS 102:15134-15139.
6) Ando et al., 2002. PNAS 99:12651-12656.
7) Horie et al., 2011. Nature 469:525-528. |