 Global Biodiversity Information Facility (GBIF) Global Biodiversity Information Facility (GBIF)
2011年GBIFは180万種10億件の生物多様性情報を提供する
国立遺伝学研究所 生命情報・DDBJ研究センター 教授 菅原秀明
|
| |
“It is the right initiative with the right goals at the right time”
“In our view, if it did not exist, it would need to be created”
(第3者によるGBIF評価から(2005)) |
|
 経緯 経緯
|
GBIFは経済協力開発機構(OECD)から始まりました。 OECDはその名称から受ける印象と異なって、 加盟国に対して科学技術政策の提言をする機能も持っています。
世紀の変わり目に、 OECDはバイオに関する科学技術政策について2つの重要な提言をしました。
一つには、メガサイエンス・フォーラムに設けられたWorking Group on Biological Informaticsの報告(1999年)に基づいて、
GBIFとGlobal Neuroinformatics Capability(GNC)の設立を提言しました。 *
もう一つは、Working Party on BiotechnologyのTask Force on Biological Resource Centres (BRC)の報告(2001年)に基づいて、 Global BRC Network (GBRCN)の設立を提言しました。**
*) http://www.gbif.org/GBIF_org/facility/OECD_Endorsement
**) http://www.wdcm.org/brc.pdf
OECDの提言の中で、 GBIF構想が最も早く実現しました。 それは、 1992年のリオ地球サミットで提案された「生物多様性条約(CBD)」を巡る議論の過程で、 各国政府が、 網羅的な生物多様性情報の必要性を認識していたからでしょう。 事実、 OECDの提言が公になった後も、 1996年OECDにWorking Groupの設置を提案した米国のみならずオランダ、 オーストラリア、 メキシコなど多数の国の熱意が続き、 2001年3月にGBIFが発足しました。 なお、 GBIFは地球規模生物多様性情報機構と訳されています。 国内では科学技術振興機構と国立環境研究所に加え、 2003年から国立遺伝学研究所を中核機関として、 東京大学大学院総合文化研究科ならびに科学博物館と連携して、 GBIF日本ノードとして活動しています。
 組織 組織
|
GBIFの組織は、理事会と執行委員会のもとに、常設の科学委員会、予算委員会、 ノード(各国に設けられるGBIF対応窓口)管理者委員会と資格審査委員会などの臨時委員会が設けられています(図1)。図1の右側にある事務局は国際入札の結果2001年に選定されたコペンハーゲン大学動物博物館で活動しています。事務局長は、OECDのWorking Group 以来米国 National Science Foundation に在籍しながら一貫してリーダシップを発揮してきたJames Edwardsが努めています。 科学委員会には4つの小委員会 DADI、DIGIT、ECAT ならびに OCB が設けられています。 |
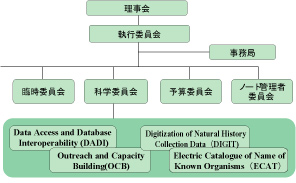
図1:GBIFの組織
|
|
- DADI - 生物多様性の課題を、多様な情報資源をオンラインで組合わせて解決するために必要なデータやメタデータなどの標準を提案します。
- DIGIT - 標本データと観測データのデジタル化を促進します。
- ECAT - 生物に関する文献や研究成果を連携するキーである学名の網羅的電子カタログをドメインごとの努力とも協調して整備します。
- OCB - ソフトウエアの提供やトレーニングによって生物多様性情報に関する技術格差を解消します。 また、 データを巡る権利関係を解きほぐします。
GBIFの運営方針は年2回開催される総会で決定されます。総会のメンバーは、 Memorandum of Understanding (MOU)にサインした47の国ならびに国に準ずる組織体と31のNGOです。 こうした組織と活動は、 26カ国の拠出金を中心に行われており、 総会ではこれら26カ国が投票権を有しています。 2006年度の予算はおよそ300万ユーロですが、 日本は米国とともにこの予算の多くを担っています。 GBIF総会が扱うこの予算に加えて、 総会メンバーがそれぞれ生物多様性情報資源と環境の充実に投資しており、 実データの殆んどは各メンバーの努力によって産み出されていることを忘れてはなりません。
|
 |
 機能 機能
|
|
2006年2月には、GBIFのデータ・ポータルサイト(http://www.gbif.net/)から、
各国に分散した159のサイトが提供する98,300種8,660万件余りのデータを、統合検索することが可能になっています。GBIFは、2003~2006年の第1期試行期間の後、2007年から第2期に入り、2011には既知の生物種を100%網羅する10億件のデータを提供することを目指しています(図2の「GBIF自ら構築する情報資源」)。
加えて、既存の文献情報、 分子生物学の情報資源、地理情報、環境情報、気象情報さらには社会経済情報
(図2の「参照する情報資源」)との連携を実現する情報環境の構築を目指しています。
この情報環境によって、 誰でも,何処でも、 何時でも、 目的にあわせて必要な情報資源を組合せて、
多様な生物の検索、 分析、 保全、 応用を進めることができるようになります。 |
図2: 生物多様性問題解決環境:Webサービスによる統合
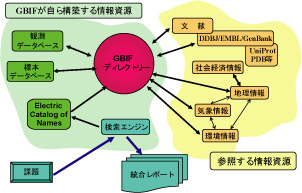
|
例えば、 特定地域での生物種の動向を、
自然史博物館に蓄積されてきた標本データ、塩基配列データ、 社会経済情報、
環境情報などを組合わせて、 予測を試みるとしましょう。 現状では、 始めに、
インターネットから必要な情報資源を探し出し、 次に、 情報資源をそれぞれ加工して、
さらに、 組合わせる工夫を凝らして初めて分析にとりかかることができます。
これに対して、 GBIFが提供する生物多様性問題解決環境が成熟してくれば、 図2にあるように基礎データを効率よく統合して入手することができます。 図2の左下からにあるように、
課題をGBIFの検索エンジンに与えると、検索エンジンが学名(Electric
Catalogue of Names)を参照した上で、 GBIFのディレクトリーを介して、標本データ、DDBJ/EMBL/GenBank (http://www.insdc.org/) の塩基配列データ、地理情報、社会経済情報および環境情報などをそれぞれ取得した上で、とりまとめて報告を戻してくれるようになります。 |
www.gbif.netがカバーしている生物界 |
|
 仮想データベースのための情報技術 仮想データベースのための情報技術
|
Webサービスと標準化
GBIFは実データがインターネットの多数のサイトに分散しているいわば仮想データベースを提供しています。 しかも、 各サイトではデータベース管理システムをはじめとしてそれぞれ多様な情報技術が使われています。 それでは、どのような情報技術によって、この仮想データベースひいては図2の問題解決環境を構築しているのでしょうか。 GBIFは、各サイトの個性をGBIFの仲間として相互理解可能なように包み込む(wrap)技術としてWebサービス(http://www.xml.nig.ac.jp/)を採用しました。 各サイトは、 GBIFが提供するツールを使って、 GBIF標準のデータ構造でGBIF標準の通信プロトコールで、 手元のデータを広く一般に提供することになります。 標準データ構造と標準プロトコールとして、現在のところ、 ノードのおよそ3分の2がDarwinCoreとDiGIR(http://digir.sourceforge.net/)を、 ヨーロッパを中心とするおよそ3分の1がABCDとBioCASE(http://www.biocase.org/)を使っています。 国立遺伝学研究所のGBIF日本ノードは国内の生物多様性情報をMySQLに格納し、 GBIFのツールを使って、 DarwinCoreとDiGIRに対応しています。
Webサービスは、 GBIFから参照する情報資源でも採用が広がっているので、 図2の左右の情報資源の連携のためにも有望な技術です。
Globally Unique Identifier (GUID)
GBIFは、学名を多様な情報資源を連携させるキーと設定してきましたが、 異なるサイトからの情報の相互参照が可能になるデータがGBIFによって増えれば増えるほど、 1件1件のデータの源である標本や菌株を特定する必要があることが明らかになってきました。 また、 学名の背景にある分類群の概念もサイト間で必ずしも共通ではありません。 そこで、 物理的実体である標本や菌株から始まって、 研究機関や、 さらに概念まで、生物に関するあらゆる対象(オブジェクト)に対して地球上で一意の識別子(GUID)を付与する枠組みの検討が始まりました。 GUIDを定義して運用できれば、 データの源である物理的実体を厳密に特定することが可能になり、 再現性がありかつ無駄な重複が無い研究の促進につながります。
GUIDの技術的基盤には、Digital Object Identifier (DOI、 http://www.doi.org/) やLife Science Identifier (LSID、 http://lsid.sourceforge.net/)といった候補があるので、 生物材料の変異や一部または全部の複製・増殖といった特徴に十分注意を払いかつGUIDの適用範囲を絞り込めば、 バイオGUIDは十分実現可能と考えられます。 またバイオGUIDの議論は国際塩基配列データベース(http://www.insdc.org/)とも協調して進んでいくことでしょう。
 関連プロジェクト Barcode of Life (BoL) 関連プロジェクト Barcode of Life (BoL)
|
GUIDは生物オブジェクトに人為的に識別子を付与する枠組みです。 これに対して、 塩基配列を生物種の識別子にしようという試みが、 Barcode of Life (http://www.barcodinglife.org/)です。 GBIF自身のプロジェクトではありませんが、 GBIFはBoLの国際コンソーシウムのメンバーとなって、相互に密接な連携を保っています。
塩基配列によるバーコード付けは、 比較的短い塩基配列によって生物種を特徴付けようとする技術です。 したがって、 特定の生物種内では共通し、 生物種間で変異が大きい部分配列を選択することになります。動物標本の場合は、 ミトコンドリアDNAのCOL1をターゲットとして配列決定とデータ蓄積が始まっています。 BoLの成果は、分類学者だけでなく生物試料を同定しようとする研究者技術者全てにとって有用な技術です。 この技術は、 特定の生物集団の専門家でなくても利用でき、 従来の手法では同定が困難か不可能であった生物試料の同定も可能とします。 バクテリアの場合は、 16sRNAの配列が分類と同定に決定的な役割を果たしており、 種のレベルでのバーコード付けはされています。 植物については、 BoLは適切な分子あるいは分子群を検討中です。
BoLのホームページによれば、2006年2月の時点でDDBJ/EMBL/GenBankに26,398件のBoLエントリーが登録され、 17,834種についてバーコードが付与されたとのことです。
 BEATLES, ”Long and winding road ” BEATLES, ”Long and winding road ”
|
GBIFの規模は一体どこまで広がるのでしょうか。 昆虫の場合は「およそ95万種が知られていている一方で、 推定800万種も未知種が生息している」と言われています(参考文献)。 したがって、 GBIFのECATにしても、 GUIDにしても、 BoLにしても全生物種を網羅するまでには遥かな道のりです。 しかし、歩みを止めなければやがては目標を達成することができ、 その時、新しい世界への扉が開かれることでしょう。
遺伝研のGBIFノードは、国内外の研究機関や研究グループと手を組んで、 一歩ずつ前進していきます。
【参考文献】
Watson, R.T., Heywood, V.H., Baste,I., Dias,B., Gamez,R., Janetos, T.Reid, W., and Ruark, G. (edts.)
“Global Biodiversity Assessment-Sumary for Policy-Makers”,
Cambridge University Press(1995) |
※
写真は菅原氏より提供していただきました。
|
